『太平洋电脑网』Folding@home志愿者数量破40万 算力超470 petaFLOPS
鉴于新冠病毒(COVID-19)在全球范围内的大规模传播 , PCMR和NVIDIA呼吁全球PC用户加入Folding@home项目贡献自己闲置的GPU计算力 , 协助抗击新冠状病毒疫情 。目前全球有超过40万志愿者参与该项目 , 拥有超过470 petaFLOPS的分布式计算能力 。
【『太平洋电脑网』Folding@home志愿者数量破40万 算力超470 petaFLOPS】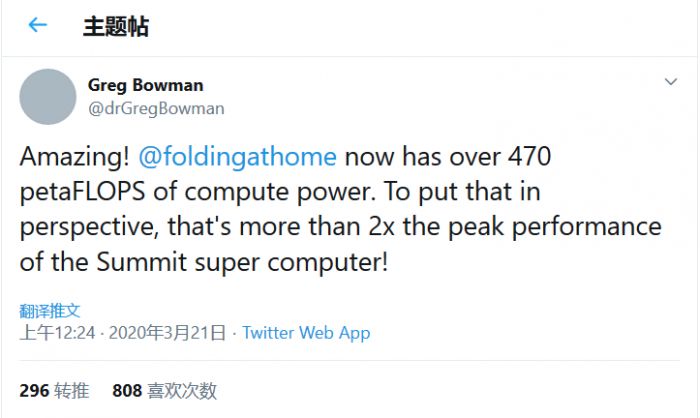
文章图片
Greg Bowman博士表示 , 在启动参与COVID-19病毒研究之前Folding@home项目的志愿者数量大约是3万人左右 , 计算性能大约是100 petaFLOPS 。 而现在该项目志愿者数量超过了40万人 , 分布式计算能力超过470 petaFLOPS 。
文章图片
如果你对这个数字并没有直观的概念 , 那么我们来做个对比 。 Summit的峰值计算为200 petaFLOPS , 而LINPACK基准测试为148.6 petaFLOPS 。 也就是说当前Folding@home项目计算能力是该超级计算机的两倍多 。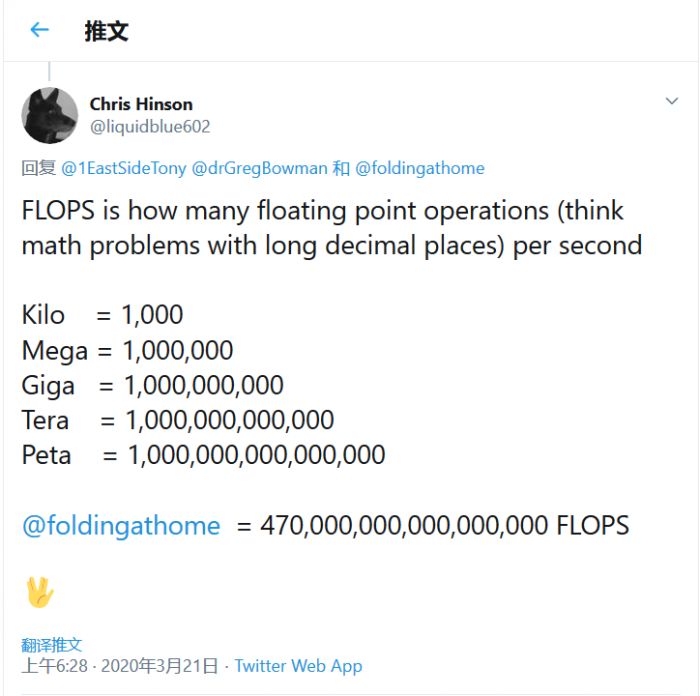
文章图片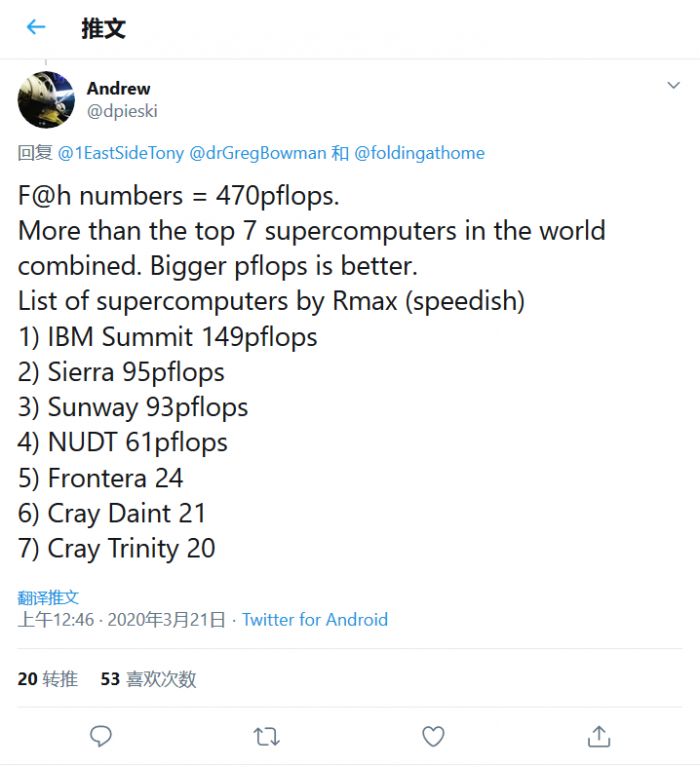
文章图片
多家媒体也参与推动了该项目的发展 , 包括Tom's Hardware, AnandTech, HotHardware和OC3D等诸多媒体也参与其中 。 此外包括EVGA等诸多硬件供应商也加入 , 技嘉还向Folding @ home研究团队捐赠了10台高端PC 。
Folding@home 指出:2019-nCoV 是 SARS 冠状病毒(SARS-CoV)的“近亲” , 并且以类似的方式起作用 。 对于两种冠状病毒 , 当病毒表面的蛋白质与肺细胞上的受体蛋白质结合时 , 肺部感染就发生了 。 这种病毒蛋白称为刺突蛋白 。
由于蛋白质不会停滞 , 会摆动 , 折叠和以多种形状展开呈现 , 所以我们不仅需要研究病毒刺突蛋白的一种形状 , 还需要研究该蛋白摆动、折叠成其他形状的所有方式 , 以便更好地了解其与 ACE2 受体是如何相互作用的 , 从而可以开发出治疗抗体 。
了解这些信息 , 需要我们对 2019-nCoV 峰值蛋白的结构进行建模 。 我们需要构建可以实现此目标的计算模型 , 但它需要大量的计算能力 。 志愿者可以 下载 Folding@home 软件, 利用闲置算力展开研究 。
【来源:cnBeta.COM】
推荐阅读
- 普通车|德系商务风对阵美式运动范 迈腾 VS 迈锐宝XL
- 普通车|选商务还是要运动? 迈腾 VS 君威
- 商家通过低保户购物而把自己的利润返还赠送低保户安装宽带或者旧电脑可行吗
- 当下电脑城/电子城/数码城的业务日益萧条,作为普通的创业者该怎样转变思路从而突围
- 汽车知识|一决高下 四款紧凑型车谁更值得买?
- 分享分享你生活中除游戏外,遇到过哪些恨电脑配置不够的情况
- 通过物理地址和IP地址能监控到啥
- 带电源的移动硬盘插电脑上就黑屏咋办
- 怎么样将家里的液晶电视和笔记本电脑连接
- 一个非专业人士适合哪款苹果电脑